消化道恶性肿瘤包括肝癌、胃癌、结直肠癌、食管癌、胰腺癌等,在全球癌症新发病例和死亡病例中分别占据23.9% 和 33.2%,疾病负担沉重[1,2]。由于消化道癌症早期常无明显临床症状,且缺乏高敏感、特异性早诊标志物,绝大多数患者确诊时已处于中晚期,错失根治性手术的最佳时机,预后不佳[3]。此外,消化道疾病的良恶性鉴别仍是临床难点,往往需要胃肠镜、CT、磁共振等多项检查进行综合判断。目前尚缺乏一种能够实现多种消化道癌种同时筛查和早期诊断的“泛癌”检测技术。近年来,基于循环肿瘤DNA的多癌种早期检测(MCED)技术快速发展,为实现消化道癌症的早期、无创、广谱检测提供了新的技术路径与临床应用可能[4]。
2025年6月5日,在樊嘉院士指导下,复旦大学附属中山医院周俭教授、杨欣荣教授团队与鹍远生物联合开展的研究成果在国际权威期刊Molecular Cancer (IF=27.7) 在线发表,论文题为“GUIDE: a prospective cohort study for blood-based early detection of gastrointestinal cancers using targeted DNA methylation and fragmentomics sequencing” [5]。本研究首次基于高通量测序平台,通过创新设计的小型靶向甲基化测序Panel,精准捕获血浆游离 DNA(cfDNA)的甲基化特征和片段组学(Fragmentomics)特征,构建出融合深度神经网络构架的多模态人工智能模型,实现对多种消化道癌症的无创检测与组织溯源(TOO)。通过首个针对消化系统癌症的前瞻性队列研究(GUIDE),研究团队系统验证了GutSeer®在消化道癌症早期检测中的卓越性能:验证集总体灵敏度为82.8%,特异性为95.8%;独立测试集总体灵敏度为81.5%,特异性为94.4%。GutSeer®不仅在检测早期消化道癌症方面表现出色,对高级别癌前病变亦具有良好识别能力,充分展示了其在特定高风险人群中的临床应用价值。值得强调的是,通过与全基因组测序(WGS)的片段组学方法进行头对头直接比较,GutSeer®的整合模型在检测性能和临床适用性方面均表现更优,突显了其作为一种实用、高效、无创的消化道癌症早期筛查和辅助诊断工具的巨大潜力。
本研究中总共招募了3,386名参与者。其中,64.0%的癌症患者被诊断为I期(534例,39.4%)和II期(334例,24.6%),为评估GutSeer®在消化道早期检测中的诊断效能提供了坚实队列基础(图1)。
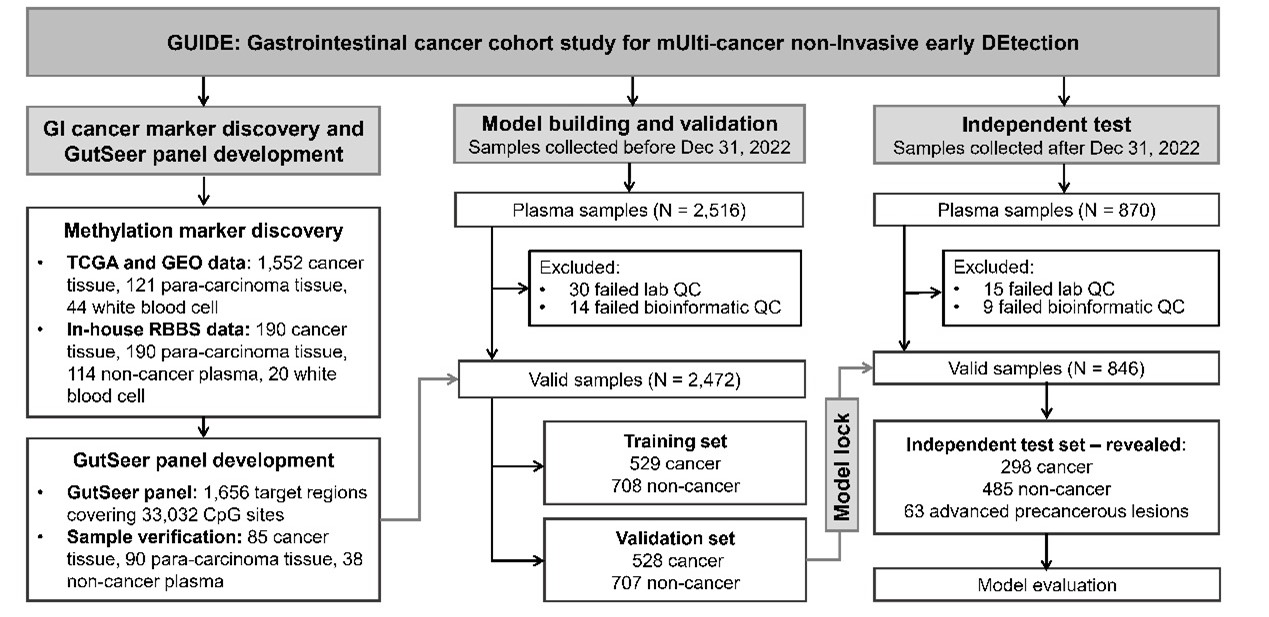 图1、GUIDE研究整体设计
图1、GUIDE研究整体设计
GutSeer®模型使用了1237个血浆样本的数据开发而成,包括529个癌症样本(63.3%为I/II期;335/529例)和年龄匹配的708个非癌症样本。在训练集中,GutSeer®模型的平均AUC为0.958,灵敏度为83.7%,特异性为95.8%(图2A)。模型的稳健性进一步在验证集中得到验证,保持了82.8%的灵敏度和95.8%的特异性,AUC为0.950(图2B)。另外,GutSeer®模型在组织溯源上表现出稳健准确性。模型在训练集中实现组织溯源准确性为83.7%,在验证集中为82.4%。准确的组织溯源性能提升了GutSeer®的临床实用性,为指导后续确诊流程和治疗计划提供了宝贵线索。
研究者探索了整合cfDNA甲基化和片段组学特征对诊断效能的提升。在癌症检测中,训练集的交叉验证结果显示,单独甲基化的模型的平均AUC为0.929,单独片段组学的模型AUC为0.934,都显著低于整合模型的0.958(图2A)。在验证集中观察到了类似的结果(图2B)。对于组织溯源也观察到了类似的准确性提升,进一步强化了结合甲基化和片段组学进行精准组织溯源的优势。
研究者进一步利用富集早期癌症病人的独立验证队列来评估GutSeer®诊断性能,队列中66.4%的癌症患者为早期患者(I/II期)。GutSeer®达到了0.921的AUC,癌症检测的灵敏度为81.5%,非癌症患者的特异性为94.4%(图2C)。该模型在组织溯源中也保持了良好性能,总体准确性为80.7%。在63名高级别癌前病变患者中,GutSeer®在8/17的结直肠癌前病变样本(灵敏度:47.1%),7/18的食管癌前病变样本(38.9%),以及6/28的胃部癌前病变样本(21.4%)中检测到癌症相关信号。这些结果突出了GutSeer®在检测早期癌症信号方面的潜力,包括高级别癌前病变,为早期干预和改善患者预后提供了希望。
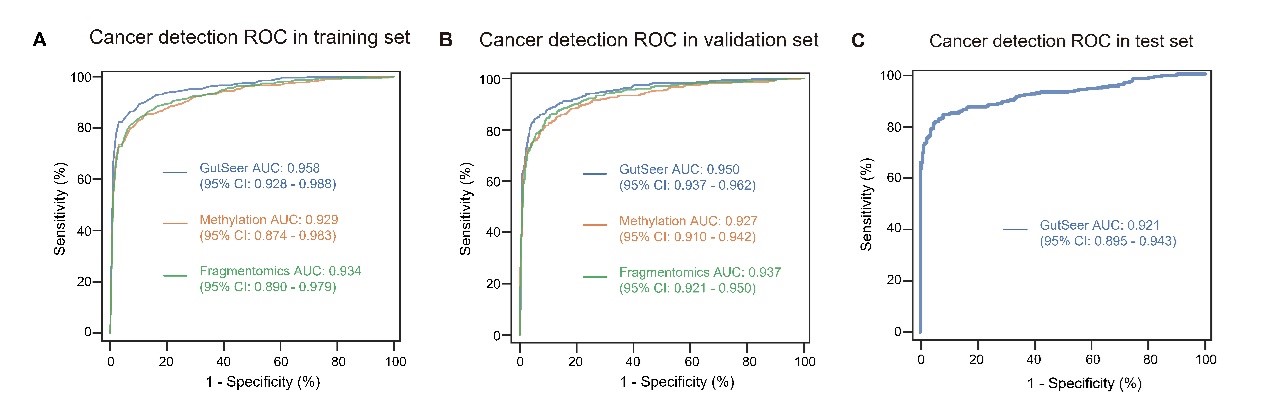 图2、评估GutSeer®消化道肿瘤检测的性能
图2、评估GutSeer®消化道肿瘤检测的性能
最后,项目组将GutSeer®和基于low-pass WGS的片段组学方法进行了头对头比较[6]。在训练和验证集中,共有1176个样本同时进行了平均测序深度为14.4X 的cfDNA WGS测序。基于同样的训练样本,项目组分别根据GutSeer®和WGS数据重新独立构建了癌症检测和组织溯源模型,并在测试集样本中进行了比较。GutSeer®在癌症检测(AUC:0.963 vs. 0.887,P < 0.001)(图3A)和组织溯源(ACC:79.6% vs. 59.0%,P < 0.001)(图3B)中均表现出更优性能。这些结果表明GutSeer®的相对较小的Panel能有效捕获全基因组片段组学特征,能同时利用多维度分子特征,以更低的成本实现优异性能。
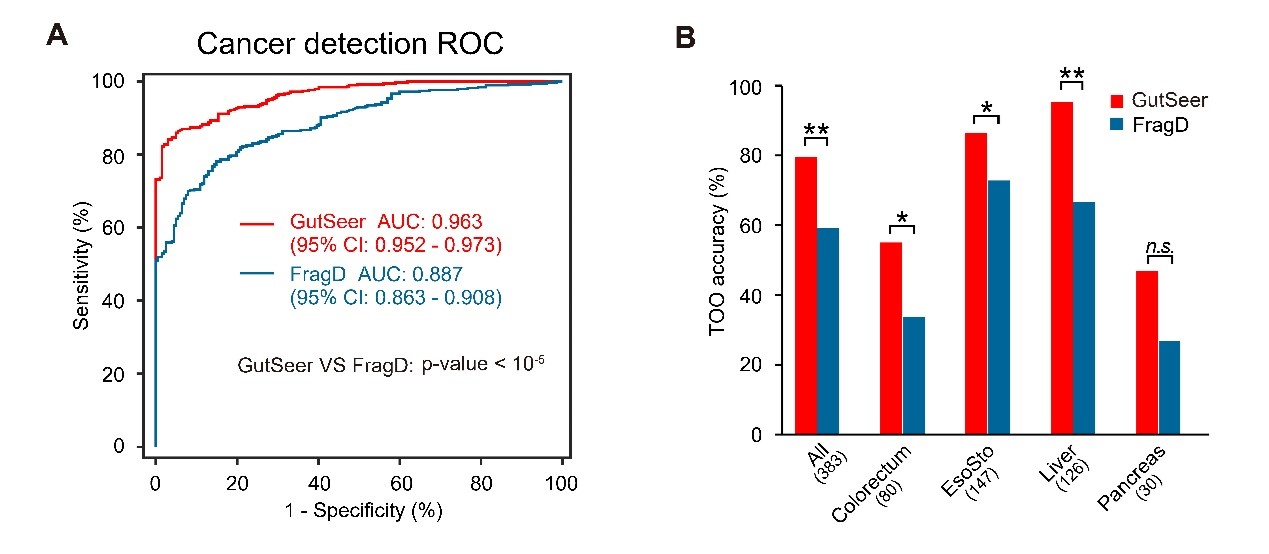 图3、GutSeer®与基于WGS方法的头对头比较
图3、GutSeer®与基于WGS方法的头对头比较
综上所述,GutSeer®方法通过整合cfDNA的甲基化模式与片段组学特征,构建包含深度神经网络构架的多模态人工智能模型,达到了检测高准确性。本项技术具有低成本、流程简便等优势,适于在临床大规模推广应用。这一研究成果不仅填补了多癌种泛癌早筛领域的技术空白,也为实现消化道癌症的“早发现、早诊断、早治疗”提供了切实可行的手段,有望显著改善患者的总体生存率与生活质量,具有广阔的临床推广前景和重大的公共卫生意义。
复旦大学附属中山医院周俭教授表示:“GutSeer®研究结果的发布,标志着我们在消化道癌症早筛领域迈出了突破性的一步。这项全球首个经过前瞻性队列验证的消化道多癌早筛技术,成功实现了对肝癌、胃癌、结直肠癌、食管癌、胰腺癌等这五大高发、高致死率消化道癌症的高精度、无创早期检测和组织溯源。 GutSeer®的成功,不仅在于其优异的性能,更在于其设计的临床导向性——它专为消化道癌症这一特定群体打造,聚焦明确的临床需求,能无缝融入现有的消化道诊疗流程,为未来大规模人群筛查奠定了坚实的基础。
作为项目课题一牵头单位,鹍远生物致力于推进该技术在国内外的产业转化,GutSeer®人类消化系统多癌基因甲基化检测试剂盒已于2024年成功获得欧盟CE认证。鹍远生物首席技术官刘蕊博士表示:“我们很荣幸与复旦大学附属中山医院等顶尖团队联合开发验证 GutSeer®技术,为全球消化道癌症防控提供早诊早筛新范式。GutSeer®前瞻性研究结果的发布,是鹍远生物在癌症早诊早筛领域深耕多年、坚持技术创新、聚焦临床实际应用的重要里程碑。这标志着基于液体活检的无创、便捷的消化道多癌早筛从概念走向现实迈出了关键一步。”
鹍远生物董事长兼首席执行官张江立先生表示:“我们将加速推进GutSeer®的产业化进程和注册步伐,积极寻求与医疗机构、体检机构、政府和支付方的广泛合作,力求让这项突破性技术早日惠及中国乃至全球的高风险人群,真正实现“早发现、早干预”,从而大幅降低消化道癌症的死亡率和疾病负担,守护人类健康。这不仅是鹍远生物的使命,也是我们对未来的承诺。”
复旦大学附属中山医院周俭教授、杨欣荣教授,长海医院金钢教授、鹍远生物刘蕊博士为该研究的共同通讯作者。复旦大学附属中山医院黄傲博士、郭德镇博士、钟芸诗教授、刘亮教授、鹍远生物苏志熙博士、湖北省肿瘤医院熊治国教授为该研究的共同第一作者。该研究为国家重点研发计划“重大慢性非传染性疾病防控研究”重点专项项目“基于液体活检技术的常见恶性肿瘤筛查及早诊技术研发与评价研究”的重要成果。
「参考文献」
[1] Bray F, Laversanne M, Sung H, Ferlay J, Siegel RL, Soerjomataram I, Jemal A: Global cancer statistics 2022: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin 2024, 74:229-263.
[2] Crosby D, Bhatia S, Brindle KM, Coussens LM, Dive C, Emberton M, Esener S, Fitzgerald RC, Gambhir SS, Kuhn P, et al: Early detection of cancer. Science 2022, 375:eaay9040.
[3] Miller KD, Nogueira L, Devasia T, Mariotto AB, Yabroff KR, Jemal A, Kramer J, Siegel RL: Cancer treatment and survivorship statistics, 2022. CA Cancer J Clin 2022, 72:409-436.
[4] Chen X, Gole J, Gore A, et al: Non-invasive early detection of cancer four years before conventional diagnosis using a blood test. Nat Commun 2020, 11:3475.
[5] Huang A, Guo D, Su Z, et al: GUIDE: A prospective cohort study for blood-based early detection of gastrointestinal cancers using targeted DNA methylation and fragmentomics sequencing. Molecular Cancer 2025, 24, 163
[6] Cristiano S, Leal A, Phallen J, Fiksel J, Adleff V, Bruhm DC, Jensen SO, Medina JE, Hruban C, White JR, et al: Genome-wide cell-free DNA fragmentation in patients with cancer. Nature 2019, 570:385-389.
* 本文仅供医学专业人士阅读参考,不涉及产品推广,非广告用途。







